Los síndromes mielodisplásicos (SMD) son trastornos hematopoyéticos clonales caracterizados por hematopoyesis ineficaz y citopenias de sangre periférica, morfología celular anormal en el examen de sangre y médula ósea y riesgo de progresión a leucemia mieloide aguda (LMA). Tienen un curso clínico variable, manifestándose en algunos pacientes como citopenias indolentes que requieren transfusiones ocasionales y en otros como enfermedades agresivas que evolucionan rápidamente a leucemias refractarias al tratamiento con un amplio espectro de gravedad en el medio (1).
Aproximadamente la mitad de los pacientes con SMD son portadores de mutaciones somáticas en los genes del espliceosoma (maquinaria celular encargada de eliminar los intrones) y, de estos, SF3B1 es el que mayormente se encuentra mutado. También se observa una gran frecuencia de mutaciones en SF3B1 en enfermedades como la leucemia linfocítica crónica, la leucemia mielomonocítica crónica, el melanoma uveal, el melanoma cutáneo y en cánceres de la mama y páncreas (2).
El gen SF3B1 codifica la subunidad 1 del factor de empalme 3b y típicamente está mutado en pacientes con SMD que presentan sideroblastos en anillo (SA). La clasificación revisada de la OMS indica que se puede hacer un diagnóstico de SMD-SA si los sideroblastos en anillo comprenden tan solo el 5% de los glóbulos rojos nucleados y hay una mutación somática de SF3B1.
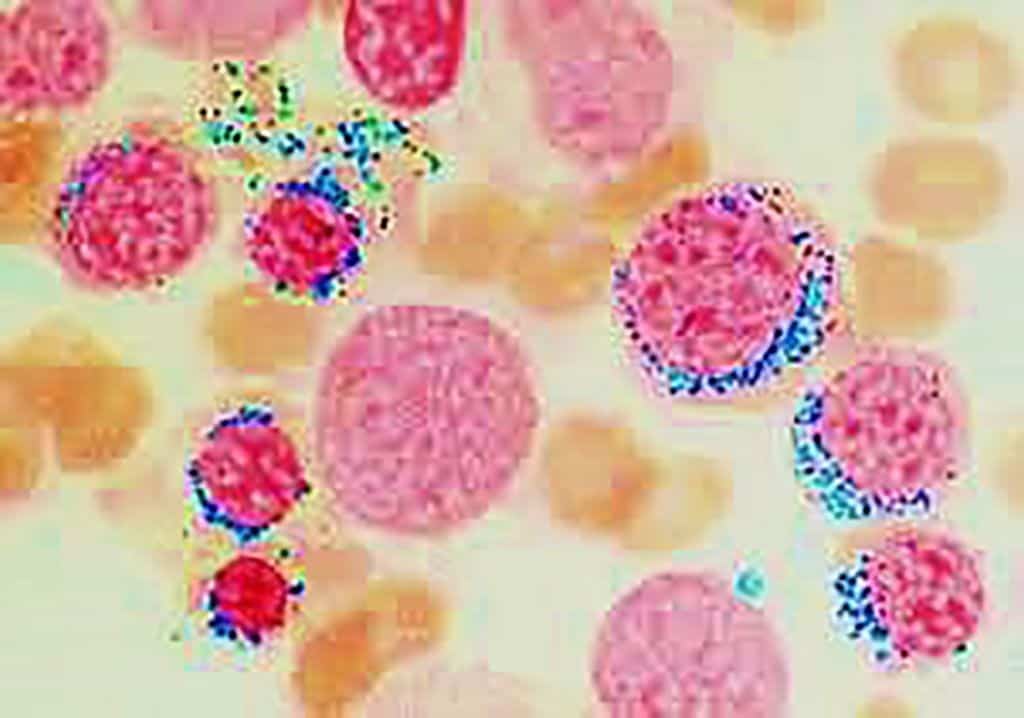
Imagen: Frotis de médula ósea de un paciente con anemia refractaria (SMD) con sideroblastos en anillo. La apariencia clásica de los sideroblastos anulares muestra la deposición de hierro en un “collar” alrededor del núcleo usando la coloración de Perls (Fotografía cortesía de la Universidad de Pavía
Varias líneas de evidencia apoyan el reconocimiento de la mutación somática de SF3B1 como marcador genético que define la enfermedad fundamentando que:
1) con mayor frecuencia representa una mutación fundacional.
2) es un determinante principal del fenotipo de la enfermedad.
3) tiene un valor pronóstico independiente de la supervivencia y el riesgo de progresión a LMA y,
4) puede predecir la respuesta a agentes específicos (3 y 4).
La mutación más comúnmente encontrada es la sustitución de una lisina por glutamato en el codón 700 (K700E), aunque pueden encontrarse otras variantes frecuentes en las inmediaciones de la misma (1). Todas estas mutaciones ocurren dentro de un grupo de 26 dominios HEAT no repetidos que se cree que están involucrados en la unión de SF3B1 a otros miembros del complejo de riboproteínas nucleares pequeñas.
La evidencia disponible indica que el SMD con SF3B1 mutado representa una entidad distinta, caracterizada principalmente por eritropoyesis ineficaz, pronóstico relativamente bueno y respuesta potencial al tratamiento con el fármaco luspatercept. Por otro lado, El SMD-SA con SF3B1 no mutado parece ser un grupo más heterogéneo con un pronóstico menos favorable y una base molecular no tan clara hasta la actualidad (3, 4).
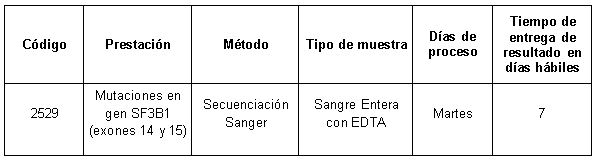
En Cibic Laboratorios contamos con la prestación Mutaciones en gen SF3B1 (exones 14 y 15). Mediante secuenciación Sanger evaluamos los exones 14 y 15 del gen SF3B1 en los cuales se observan las mutaciones más frecuentes.
Prestación disponible en Cibic Laboratorios:
Para conocer las condiciones del paciente, de almacenamiento y de envío de la muestra y otros datos sobre las prácticas consulte al manual de prestaciones y a la extranet.
Referencias
1) Christopher J. Gibson y cols. Chapter 60 – Myelodysplastic Syndromes, Hematology (Seventh Edition), Elsevier, 2018, Pages 944-969.
2) Rachel B. Darman y cols. Cancer-Associated SF3B1 Hotspot Mutations Induce Cryptic 3′ Splice Site Selection through Use of a Different Branch Point, Cell Reports, Volume 13, Issue 5, 2015, Pages 1033-1045.
3) Arber DA, Orazi A, Hasserjian R, et al. The 2016 revision to the World Health Organization classification of myeloid neoplasms and acute leukemia. Blood. 2016;127(20): 2391-2405.
4) Malcovati L, y cols. SF3B1-mutant MDS as a distinct disease subtype: a proposal from the International Working Group for the Prognosis of MDS. Blood. 2020 Jul 9;136(2):157-170.
Para mayor información o consultas:
 Sección: Biología Molecular
Sección: Biología Molecular
Lic. Alan Gomez.
Tel 0341-486-1600. Interno: 243/225
 Sección: Biología Molecular
Sección: Biología Molecular
Lic. Analía Seravalle.
Tel 0341-486-1600. Interno: 242.

