El Microarray Cromosómico o CMA (del inglés, Chromosomal Microarray) es una técnica que detecta variantes en el número de copias o CNVs (del inglés, Copy Number Variations) en el ADN, que se generan a partir de diversos rearreglos cromosómicos constitutivos que tienen lugar durante la meiosis. El CMA permite identificar anomalías cromosómicas desbalanceadas que están asociados a diversos desórdenes genéticos que no son detectados por cariotipo convencional debido a su limitada resolución y operador dependencia.
La utilización del microarray cromosómico o CMA está recomendada por las distintas guías internacionales como primera línea diagnóstica en pacientes con retraso madurativo/ discapacidad intelectual, trastorno del espectro autista y/o múltiples anomalías congénitas. El uso del cariotipo convencional por bandeo G se orientaría en primera instancia a pacientes con fenotipo evidente de síndrome genético conocido (ej.: síndrome de Down), pacientes con historia familiar de re-arreglos cromosómicos y en pacientes con antecedentes de múltiples pérdidas de embarazos.
Esta metodología cuenta con una resolución media de entre 100-200kb a lo largo de todo el genoma y de entre 30-50kb en regiones de interés. CMA permite detectar grandes y pequeñas variaciones en el número de copias (CNVs), también conocidas como microdeleciones/ microduplicaciones, que no son posibles de detectar con el cariotipo convencional. Distintos estudios han demostrado que el aumento del rédito diagnóstico en este grupo de pacientes es del 15-20% en relación con el cariotipo. Si bien el CMA no puede detectar re-arreglos balanceados (sin pérdida ni ganancia de material genético) ni mosaicismo en bajo porcentaje, este tipo de anomalías son relativamente infrecuentes en este grupo de pacientes (<1%).
Actualmente en Héritas, empresa de genómica asociada a Cibic Laboratorios, se utiliza la tecnología de Illumina para microarray cromosómico basada en sondas de SNPs. Las mismas están distribuidas a lo largo de todo el genoma, con mayor representación de éstas en zonas de interés (regiones ISCA: catálogo internacional de aberraciones cromosómicas). En función de la distribución alélica (B-allele frequency) y la intensidad de la señal (log R ratio) de las mismas, se puede inferir el número de copias de las distintas regiones representadas en el array. Las ventajas que presenta la tecnología de SNPs (Illumina) es su mayor sensibilidad en la detección de mosaicos (aproximadamente 5%) y de disomía uniparental (isodisomías, de aproximadamente 3 Mb).
Prestación disponible en Cibic Laboratorios:
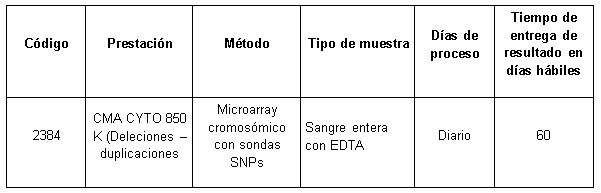
Para conocer las condiciones del paciente, de almacenamiento y de envío de la muestra y otros datos sobre las prácticas consulte al manual de prestaciones y a la extranet.
Para la clasificación e interpretación de las variantes halladas se siguen los lineamientos del American College of Medical Genetics and Genomics (ACMG) teniendo en cuenta principalmente el tamaño de la variante, la localización y naturaleza (ganancia o pérdida)
Referencias:
– Miller y cols., 2010. Consensus Statement: Chromosomal Microarray Is a First-Tier Clinical Diagnostic Test for Individuals with Developmental Disabilities or Congenital Anomalies. Am J of Hum Gen 86, 749–764.
– Kearney HM, Thorland EC, Brown KK, Quintero-Rivera F, South ST, Working Group of the American College of Medical Genetics Laboratory Quality Assurance Committee. American College of Medical Genetics standards and guidelines for interpretation and reporting of postnatal constitutional copy number variants. Genet Med 2011;13:680-5.
Para mayor información o consultas:
 Sección: Biología Molecular
Sección: Biología Molecular
Licenciado Alan Gómez
Tel: 0341-4722424, internos: 243/ 225

