La subfamilia de proteínas RAS (KRAS, HRAS y NRAS) son pequeñas GTPasas que actúan como reguladores moleculares, controlando un amplio espectro de actividades celulares, como la proliferación y la supervivencia celular (1).
Las tres proteínas RAS comparten una alta homología de secuencia, excepto por la región hipervariable del extremo C, que se cree que confiere la función específica de cada proteína. Se las consideran interruptores moleculares porque ciclan entre las conformaciones de “encendido” y “apagado”, que son dadas por la unión de GTP y GDP, respectivamente. La transición entre ambos estados está regulada por dos familias de proteínas diferentes. Los factores de intercambio de nucleótidos de guanina (GEF) promueven la disociación de GDP y la unión de GTP, mientras que las proteínas activadoras de GTPasa (GAP) estimulan la actividad GTPasa intrínseca de RAS para apagar esta señal (Fig1).
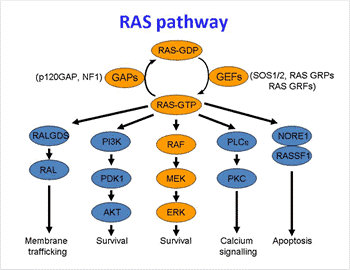
Fig1. Esquema de la vía de señalización RAS/MAPK
Se ha reportado que hasta un tercio de los cánceres humanos son portadores de mutaciones sin sentido de ganancia de función que ocurren dentro de una región conservada dentro de las diferentes proteínas RAS. KRAS es la proteína que se encuentra mutada con mayor frecuencia, seguida de NRAS y HRAS. Se han descrito cuarenta y cuatro mutaciones puntuales diferentes y el 99,2% de ellas están ubicadas en los codones 12, 13 y 61 (1). Todas estas mutaciones canónicas provocan la pérdida de la actividad GTPasa intrínseca y/o estimulada por GAP de las proteínas RAS, lo que lleva a una forma de RAS activada constitutivamente.
Curiosamente, la isoforma que mayormente se encuentra mutada, así como la posición del codón y la sustitución de aminoácidos varía entre las proteínas RAS en los diferentes cánceres humanos, pero aún no se conoce la razón de esto. Las alteraciones oncogénicas en KRAS son más frecuentes en pacientes con carcinoma de páncreas, tumores colorrectales y neoplasias pulmonares. Por su lado, las mutaciones en HRAS se pueden encontrar en neoplasias malignas dermatológicas y cánceres de cabeza y cuello, mientras que las mutaciones en NRAS son comunes en melanomas y en algunas neoplasias malignas hematopoyéticas (Fig 2) (1).
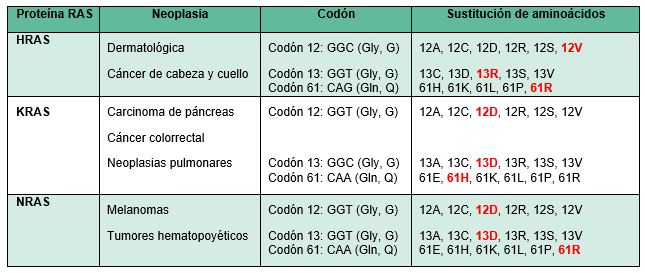
Fig2. Mutaciones más comunes en los codones individuales de las proteínas RAS. Resaltado en rojo las observadas con mayor frecuencia. Gly y G: glicina; Gln y Q: glutamina; A: alanina; C: cisteína; D: ácido aspártico; R: arginina; S: serina; V: valina; H: histidina; K: lisina; L: leucina; P: prolina; E: ácido glutámico. Tomado y adaptado de Muñoz-Maldonado et al. 2019.
En la práctica clínica, evaluar en el tumor el estatus de los genes que codifican las proteínas RAS, principalmente en los codones frecuentemente mutados (12, 13 y 61), tiene demostrada utilidad como marcadores pronósticos, predictivos de respuesta al tratamiento y recientemente para la selección de pacientes candidatos a recibir terapia dirigida (2, 3).
Así es que, por ejemplo, la National Comprehensive Cancer Network (NCCN) (4) y la American Society of Clinical Oncologists (ASCO) recomiendan que todos los pacientes diagnosticados con cáncer colorrectal metastásico en estadio IV y aquellos que tienen una recurrencia deben someterse a una prueba de mutación NRAS y KRAS, además de todos aquellos pacientes que están por comenzar terapia anti-EGFR, ya que una mutación en alguno de estos genes es un biomarcador predictivo negativo de una respuesta deficiente a los tratamientos anti-EGFR como cetuximab o panitumumab.
Por otro lado, en este último tiempo se han estado desarrollando terapias dirigidas a mutaciones específicas, principalmente en el gen KRAS (2) . Recientemente ha sido aprobada por organismos reguladores como FDA (6) y ANMAT (bajo régimen especial) (7) la droga sotorasib para tratar a pacientes con cáncer de pulmón de células no pequeñas (CPCNP) localmente avanzado o metastásico que ya hayan recibido al menos un tratamiento sistémico y sean portadores de la mutación G12C en el gen KRAS.
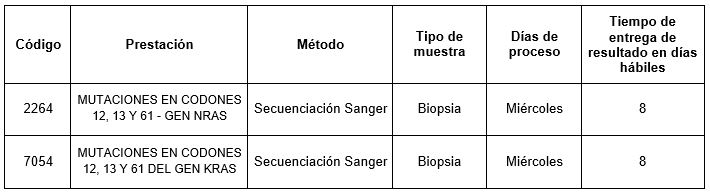
En Cibic Laboratorios, contamos con las siguientes determinaciones que nos permiten detectar mutaciones en los codones 12, 13 y 61 de los genes KRAS y NRAS mediante secuenciación Sanger.
Prestaciones disponibles en Cibic:
Para conocer las condiciones del paciente, de almacenamiento y de envío de la muestra y otros datos sobre las prácticas consulte al manual de prestaciones y a la extranet.
Referencias
1. Muñoz-Maldonado y cols. Comparative Analysis of Individual RAS Mutations in Cancer Biology. Front Oncol. 2019 Oct 18;9:1088. doi: 10.3389/fonc.2019.01088. PMID: 31681616; PMCID: PMC6813200.
2. Punekar, S.R., y cols. The current state of the art and future trends in RAS-targeted cancer therapies. Nat Rev Clin Oncol 19, 637–655 (2022). https://doi.org/10.1038/s41571-022-00671-9.
3. Schirripa M. y cols. A. Role of NRAS mutations as prognostic and predictive markers in metastatic colorectal cancer. Int J Cancer. 2015 Jan 1;136(1):83-90. doi: 10.1002/ijc.28955. Epub 2014 May 28. PMID: 24806288.
4. https://www.nccn.org/professionals/physician_gls/pdf/colon.pdf
5. https://ascopubs.org/doi/full/10.1200/JCO.22.01690
6. https://www.fda.gov/drugs/resources-information-approved-drugs/fda-grants-accelerated-approval sotorasib-kras-g12c-mutated-nsclc
7. https://www.argentina.gob.ar/anmat/regulados/medicamentos/registros-especiales
Para mayor información o consultas:

Sección: Biología Molecular
Lic. Alan Gomez.
Tel 0341-4722424. Interno: 225

Sección: Biología Molecular
Lic. Analía Seravalle.
Tel 0341-4722424. Interno: 242.

