Las leucemias mieloides agudas (LMAs) representan un grupo de neoplasias mieloides con marcada heterogeneidad genética y etiología diversa. Resultan de una proliferación clonal de células precursoras hematopoyéticas anormales con diferentes grados de diferenciación, que infiltran la médula ósea y en ocasiones, otros órganos. Representan el 15 a 20% de las leucemias agudas en niños y adolescentes, y hasta el 80% en adultos.
Clasificación de leucemias agudas (OMS revisión 2016) (1)
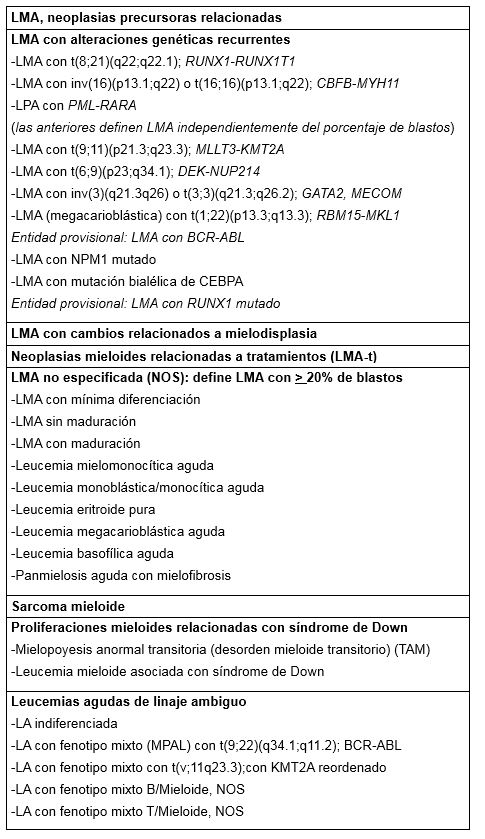
El diagnóstico y clasificación, como así también el pronóstico y seguimiento de LMA, incluyen estudios de citometría de flujo y citogenéticos-moleculares. El procedimiento de rutina para la realización de estos estudios es el aspirado de médula ósea. En pacientes hiperleucocitarios, estas determinaciones pueden realizarse en sangre periférica.
Estudios de citometría de flujo
El inmunofenotipo por citometría de flujo es fundamental para determinar las líneas involucradas en el clon leucémico e identificar patrones de expresión antigénica anómalos que serán útiles para cuantificar la enfermedad residual mínima (ERM), (Tabla 1 y 2) (2).
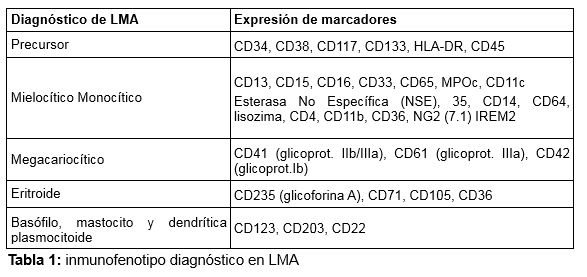
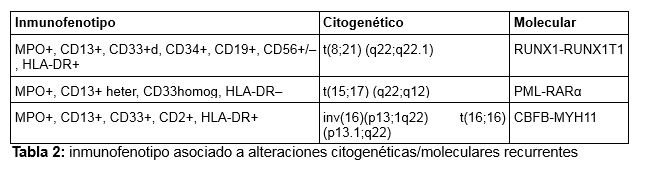
Estudio citogenético – molecular
El estudio citogenético convencional (bandeo G) es mandatorio en la evaluación diagnóstica de LMA, permitiendo su clasificación y definiendo subgrupos de riesgo ya que tiene un peso de valor pronóstico independiente. Se deben analizar un mínimo de 20 metafases para definir un cariotipo normal. Aproximadamente el 55% de los pacientes presentan alteraciones citogenéticas, y hasta 80-85% en los niños (3).
El estudio molecular por FISH o RT-PCR es una herramienta útil para evidenciar alteraciones crípticas, y cuando el estudio citogenético no es concluyente.
Mediante técnicas moleculares se pueden detectar las siguientes alteraciones: PML-RARA, RUNX1- RUNX1T1, CBFB-MYH11, MLLT3-KMT2A, NPM1, CEBPA, FLT3, KIT, RUNX1, ASXL1, TP53 y BCR-ABL (4).
Factores pronósticos
El cariotipo y determinadas alteraciones moleculares son los factores pronósticos más importantes para predecir la probabilidad de alcanzar una remisión completa, riesgo de recaída y supervivencia global.
Según ELN (European Leukemia Network) se definen tres grupos:
• Favorable:
◦ t(8;21)(q22;q22.1); RUNX1-RUNX1T1
◦ inv(16)(p13.1q22) o t(16;16)(p13.1;q22);CBFB-MYH11
◦ NPM1 mutado in FLT3-ITD/FLT3-ITD BAJO
◦ CEBPA mutación bialélica
• Intermedio:
◦ NMP1 mutado y FLT3-ITD (alto)
◦ NPM1 no mutado sin FLT3-ITD/FLT3-ITD bajo (sin alt. genéticas de riesgo adverso) t(9;11)(p21.3;q23.3); MLLT3-KMT2A
◦ Alteraciones citogenéticas no clasificadas como favorables o desfavorables
• Adverso:
◦ inv(3)(q21.3q26.2);t(3;3)(q21.3;q26.2);GATA2, MECOM
◦ t(6;9)(p23;q34.1); DEK-NUP214
◦ t(v;11)(v;q23.3); KMT2A (MLL) reordenado
◦ t(9;22)(q34.1;q11.2); BCR-ABL
◦ -5 o del(5q); -7; -17/alt(17p)
◦ cariotipo complejo, cariotipo monosomal
◦ NMP1 no mutado y FLT3-ITD (alto)
◦ RUNX1 mutado
◦ ASXL mutado
◦ TP53 mutado
En relación a los estudios citogenéticos, el cariotipo monosómico, que implica la presencia de al menos dos monosomías autosómicas o una monosomía autosómica en combinación con una alteración estructural (excepto alteraciones en el CBF), se asocia a riesgo adverso. Los cariotipos hiperploides puros (ganancia de cromosomas completos >65, sin alteraciones estructurales ni monosomías asociadas) tienen especialmente un mal pronóstico.
En relación a los estudios moleculares, los pacientes con ratio <0.5 (bajo) (relación entre cantidad de alelo portador de ITD y de alelo salvaje) tienen un riesgo comparable a los pacientes NPM1 mutado sin FLT3-ITD, estando dentro del grupo de riesgo favorable. Se debe considerar también la evaluación de mutaciones en los codones D835 e I836 del dominio quinasa de FLT3, ya que podrían representar blancos terapéuticos. La presencia del rearreglo BCR-ABL incluye a la LMA en el grupo de pronostico adverso (4).
Prestaciones disponibles en Cibic:
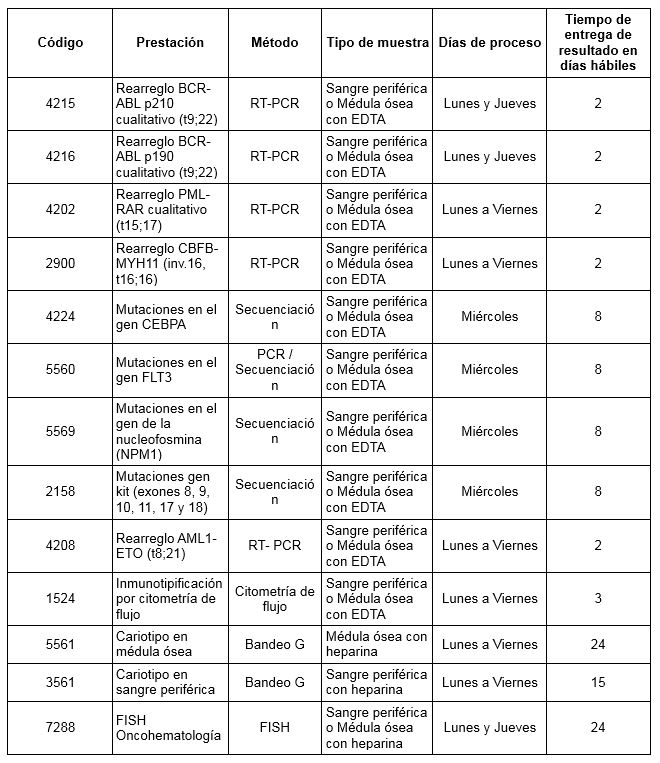
Para conocer las condiciones del paciente, de almacenamiento y de envío de la muestra y otros datos sobre las prácticas consulte al manual de prestaciones y a la extranet.
Referencias
1. Aber D, Orazi A, Hasserjian R et al. The 2016 revision of the World Health Organization classification of myeloid neoplasms and acute leukemia. Blood 2016; 127: 2391-2405.
2. Béné MC, Nebe T, Bettelheim P, et al. Immunophenotyping of acute leukemia and lymphoproliferative disorders: a consensus proposal of the European Leukemia Net Work Package 10. Leukemia 2011; 25(4):567-574.
3. Grimwade D. et al. Refinement of cytogenetic classification in acute myeloid leukemia: determination of prognostic significance of rare recurring chromosomal abnormalities among 5876 younger adult patients treated in the United Kingdom Medical Research Council trials. Blood. 2010; 116(3):354-65.
4. Döhner H, Estey EH, Grimwade D et al. Diagnosis and management of AML in adults: 2017 ENL recommendations from an international expert panel. Blood 2016; 129:424-447.
Para mayor información o consultas:
Sección: Biología Molecular

Lic. Analía Seravalle
Tel: 0341-4499444. Int: 242
Sección: Citometría de Flujo

Bioq. María Victoria Schoepf
Tel: 0341-4499444. Int: 259
Sección: Citogenética

Dr. Carlos Zumoffen
Tel: 0341-4499444. Int: 283
