Autores: Dr. Rodolfo Notario y Dra. Noemí Borda
Las bacterias se hacen rápidamente resistentes a los antibióticos, en parte por su rápida capacidad de adaptación y en parte por el mal uso que hacemos de esas drogas, a nivel de medicina humana, veterinaria y la automedicación. Han surgido cepas multirresistentes tanto en los pacientes internados como en la comunidad, particularmente de los géneros Klebsiella y otras enterobacterias, Pseudomonas, Acinetobacter, Staphylococcus y Enterococcus.
Frente al ambiente hostil que generan los antimicrobianos, las bacterias se adaptan rápidamente por medio de mutaciones, las cuales pueden transferir a otras bacterias de distintas maneras. Esas mutaciones pueden determinar varios mecanismos que conducen a la resistencia. Los trabajos sobre el tema suelen ser complejos y poco prácticos para el clínico. Resumimos aquí, el origen de la resistencia, la trasferencia y los distintos mecanismos involucrados.
1. Origen de la resistencia a los antimicrobianos (ATM). En toda población de seres vivos hay mutantes. Así se explica que, tan pronto se presenta un nuevo ATM pueda suceder que aparezcan cepas resistentes.
Mutación. Es un cambio en el genotipo que se produce durante la copia del ADN. Es al azar, espontáneo y una vez producido es heredable. La mutación se produce con una frecuencia de 1 cada 104 a 1010. Cuando se efectúan tratamientos combinados de dos ATM simultáneos, la posibilidad de aparición de una cepa con la doble mutación es mucho más baja lo que avala los tratamientos combinados. La mutación se ubica en genes o en conjuntos de genes, que pueden estar en el cromosoma, en un plásmido, codificando para solo un ATM o un grupo de ellos formando “cassettes”, pudiendo encontrarse en elementos genéticos móviles como transposones o integrones.
Selección de mutantes: Al efectuar un tratamiento ATM se pueden eliminar las bacterias sensibles del sitio de la infección o de la flora del paciente y seleccionarse cepas resistentes. Ejemplo: una persona que recibió ATM puede ser portadora de cepas resistentes. A través de toallas, lavatorios, estetoscopios, tensiómetros, teléfonos o simplemente a través de las manos, estas cepas se pueden difundir en el sanatorio u hospital
2. Transmisión de la resistencia. El gen de resistencia puede ser trasmitido de una bacteria a otra por plásmidos, por bacteriófagos o por transformación.
• Transformación: algunas bacterias son capaces de tomar ADN exógeno del medio (incluso proveniente de microorganismos muertos).
• Transducción: un fago lisogénico, estando en estado de profago (incorporado en el ADN cromosómico bacteriano) toma parte del cromosoma, se replica y lleva esa información genética a nuevas células bacterianas.
• Conjugación: es la transferencia de material genético por contacto de una bacteria con otra para lo cual debe existir un plásmido transmisible en una de ellas. Plásmido es ADN extracromosómico. Este plásmido contiene un factor de transferencia (FTR) o factor sexual. Este FTR codifica para la elaboración de una proteína llamada pelo sexual que produce un anclaje entre la bacteria donante llamada F+ y la receptora llamada F- y permite el pasaje. Existen diferentes mecanismos de conjugación. Una bacteria con un plásmido puede tener varias copias del mismo y puede tener numerosos plásmidos diferentes. Al pasarle a otra un plásmido, éste puede contener información de resistencia a varios grupos de ATM simultáneos.
Así, una persona ingresa a un sanatorio infectada con una bacteria sensible. Una bacteria resistente de otro paciente, puede transferirle genes de resistencia.
3. Mecanismos de resistencia.
A través de la mutación o de la trasmisión de genes se adquiere, entonces, la capacidad de ser resistente a un ATM. Ahora bien, qué es lo que cambia en la bacteria para hacerse resistente, o sea ¿cuál es el mecanismo íntimo de la resistencia?.
Los más conocidos son:
• Producción de enzimas inactivantes: por ejemplo, beta lactamasas frente a los ATM beta lactámicos. Existen numerosísimas beta lactamasas, desde las penicilasas a las carbapenemasas. Otros ATM pueden ser inactivados por enzimas, como el cloranfenicol, los aminoglucósidos (por ej: las enzimas adenilasas ANT (3´´), ANT (2´´) y fosforilasa APH (3´). La resistencia de Staphylococcus aureus a macrólidos se efectúa por metilasas codificadas en genes erm y bombas de expulsión.
• Cambio en el sitio de acción del ATM.
La bacteria elabora un sitio con menos afinidad por el ATM. Por ejemplo cambio en las proteínas ligadoras de penicilinas (PLP) o sitio de acción de las penicilinas y demás beta lactámicos.
• Impermeabilidad.
Por ejemplo, la vancomicina no puede ingresar a la pared de las bacterias gram negativas. En Pseudomonas aeruginosa la pérdida de la porina OprD puede impedir el ingreso de imipenem y causar resistencia a ese fármaco.
• Bombas de expulsión (eflujo).
La bacteria tiene bomba de expulsión para expulsar metabolitos, entre ellos, los ATM. De esta manera se puede manifestar resistencia simultánea a varias familias de ATM, como es el caso de la bomba de expulsión MexXY-OprM en Pseudomonas aeruginosa.
A veces se presentan en una misma bacteria varios de los mecanismos mencionados que las hacen resistentes a varios grupos de ATM.
4. Prueba de sensibilidad a los antimicrobianos (PSA)
También llamada antibiograma consiste en enfrentar in vitro uno o varios ATM con la bacteria causante de una infección. Pueden ser por difusión o cuantitativas.
Por difusión se puede saber, siguiendo las recomendaciones internacionales, la sensibilidad a un grupo de ATM y además confirmar fenotípicamente la presencia de enzimas como beta lactamasas, incluyendo carbapenemasas, lo que debe confirmarse genotípicamente por biología molecular.
Las pruebas cuantitativas son imprescindibles para agentes como los glucopéptidos, carbapenemes y polimixinas. Son además necesarias para determinar los parámetros farmacocinéticos y farmacodinámicos (PK-PD), teniendo muy en cuenta factores del paciente que pueden alterar la concentración del antimicrobiano en el foco de infección, como 4º espacio, diálisis, insuficiencia renal, etc.
Estamos viviendo una época en la que las bacterias han adquirido alarmante nivel de resistencia. Se denomina multirresistente a la bacteria resistente a agentes de 3 o más grupos de ATM, extremadamente resistente a la bacteria resistente a agentes de todos los grupos exepto dos y panresistente a la bacteria resistente a agentes de todos los grupos de ATM.
En los casos de infecciones graves, la mortalidad está en relación a la demora del inicio del tratamiento adecuado. Es vital el inicio del tratamiento específico en las primeras horas.
Los datos de resistencia de las bacterias deben registrarse en cada nosocomio y establecer las bases del tratamiento empírico. Una vez obtenido la sensibilidad del microorganismo por el antibiograma, se puede de-escalar, es decir, cambiar el ATM empírico por otro sensible en el antibiograma a los efectos de evitar la presión de selección de resistencia. Por ejemplo, si se había indicado en las primera horas un carbapenem y cuando se tienen los datos del antibiograma se ve que la bacteria es sensible a la ampicilina con sulbactam, se cambia el tratamiento con carbapenem por este otro beta lactámico para evitar la presión de selección de cepas con carbapenemasas.
Para determinar la sensibilidad y resistencia de las diferentes bacterias se puede efectuar el antibiograma convencional por discos según las normas de CLSI/EUCAST, establecer por método enzimático la presencia de beta lactamasa de espectro extendido (BLEE), y de metilasa frente a macrólidos y lincosaminas, determinar la CIM a varios ATB por método automatizado e investigar los genes MecA y el que codifica para la carbapenemasa KPC.
Con la ayuda de las pruebas in vitro el clínico puede indicar adecuadamente ATM y efectuar el seguimiento del tratamiento.
Prestaciones disponibles en Cibic:
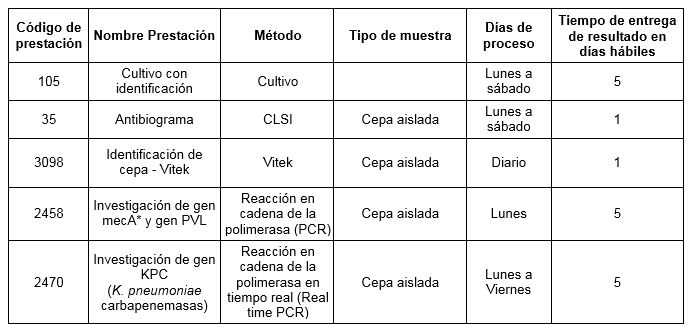
* Detecta la presencia del cassette cromosómico estafilocócico SCCmec tipo IV que contiene al gen mecA
Para conocer las condiciones del paciente, de almacenamiento y de envío de la muestra y otros datos sobre las prácticas consulte al manual de prestaciones y a la extranet.
Bibliografía
1- Perez F and Van Duin D. Carapenem-resistaant Enterobacteriaceae: a menace to our most vulnerable patients. Cleveland Clinica J of Medicine 2013;80:225-33
2- Kumarn A et al. Duration of hypotension before initiation of effective antimicrobial therapy is the critical determinant of survival in human septic shock. Crit Care Med 2006;34:1589-96.
3- Livermore D et al. What remains against carbapenem-resistant Enterobacteriaceae? Evaluation of chloramphenicol, ciprofloxacin, colistin, fosfomycin, minocycline, nitrofurantoin, temocillin and tigecycline. Int J Antimicrob Agents 2011;37:415-19
4- Notario R. Microbiología para el Médico 2ª Ed. UNR Ed. Rosario, 2014
Leon Gil C et al. Documento de Consenso (SEMES-SEMICYUC). Recomendaciones del manejo diagnóstico-terapéutico inicial y multidisciplinario de la sepsis grave en los servicio s de urgencias hospitalarios. Med Intensiva 2007;1:375-87
5- Casellas JM, Nannini E, Radice M, Cocconi E, Lejona S, Borda N, Peres, Tomé G, Notario R, Truscello M, Lovesio C, Gutkind G, Fay F. Estudio de un brote debido a aislados de Klebsiella pneumoniae productores de betalactamasas de espectro extendido en un centro asistencial de Rosario – Argentina. Rev Panam Infectol 2005, 7:21-7
6- Magiorakos AP et al. Multidrug-resistant, extensively drug-resistant, and pandrug-resistant bacteria: an international expert proposal for interim standard definition for acquired resistance. Clin Microbiol Infect 2012;18:268-81
Para mayor información o consultas:
Dr. Noemí Borda, Responsable de Microbiología.
Tel: 0341 – 4499444. Int: 228.

